Acide ribonucléique
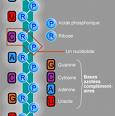
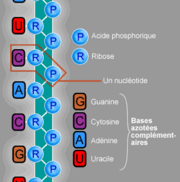
L'Acide ribonucléique ou ARN est un polymère linéaire constitué d'un enchaînement de nucléotides. Chaque nucléotide contient un groupement phosphate, un sucre, le ribose et une base azotée.
Recherche sur Google Images :
Source image : www.colvir.net Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Définitions :
- Polymère d'acide organique composé de nucléotides tels que l'adénosine, la guanosine, la cytidine et l'uridine.... (source : biofondations.gc)
- autre molécule géante complexe polymère de nucléotides, support de l'information génétique, soit chez certains virus soit apparaissant... (source : iufm.unice)
| Acides nucléiques |
|---|
L'Acide ribonucléique ou ARN est un polymère linéaire constitué d'un enchaînement de nucléotides. Chaque nucléotide contient un groupement phosphate, un sucre, le ribose et une base azotée. Les nucléotides sont liés les uns aux autres par des liaisons phosphodiester. On trouve quatre bases azotées dans l'ARN, l'adénine, la guanine, la cytidine et l'uracile. L'ARN a de nombreuses similarités avec l'ADN, avec cependant quelques différences importantes : sur le plan de la structure, l'ARN contient un ribose à la place du désoxyribose de l'ADN, ce qui rend l'ARN chimiquement plus instable et la thymine de l'ADN y est remplacée par l'uracile, qui possède les même propriétés d'appariement de base avec l'adénine. Sur le plan fonctionnel, l'ARN est le plus fréquemment trouvé dans les cellules sous forme de simple brin, alors que l'ADN est présent sous forme de deux brins complémentaires, formant une double hélice. Enfin les molécules d'ARN trouvées dans les cellules sont plus courtes (de quelques dizaines à quelques milliers de nucléotides) que l'ADN du génome (de quelques millions à quelques milliards de nucléotides.
Dans la cellule, l'ARN est produit par à partir de l'ADN localisé dans le noyau. L'ARN est par conséquent une copie d'une région de l'un des brins de l'ADN. Les enzymes qui effectuent cette copie ADN->ARN se nomment des ARN polymérases. Les ARN ainsi produits peuvent avoir trois grands types de fonctions, ils peuvent être support de l'information génétique d'un ou plusieurs gènes codant pour des protéines (on parle alors d'ARN messagers), ils peuvent adopter une structure secondaire et tertiaire stable et accomplir des fonctions catalytiques (par exemple l'ARN ribosomique), ils peuvent enfin servir de guide ou de matrice pour des fonctions catalytiques accomplies par des facteurs protéiques (par exemple les microARN)
Structure de l'ARN
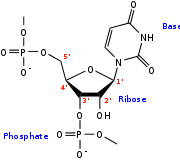
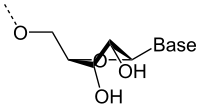
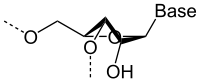
L'ARN est un acide nucléique autrement dit une molécule constituée d'un enchainement de nucléotides. Chaque nucléotide de l'ARN est constitué d'un pentose, le ribose, dont les atomes de carbone sont numérotés de 1'à 5', d'une base azotée et d'un groupement phosphate. La base azotée est reliée par un atome d'azote au carbone 1'du ribose. Les nucléotides sont liés les uns aux autres par des groupements phosphate, par l'intermédiaire de liaisons phosphodiester au niveau des carbones 3'et 5'. L'ARN possède quatre bases azotées différentes : l'adénine (notée A), l'uracile (noté U), la cytosine (notée C) et la guanine (notée G). La thymine de l'ADN est remplacée par l'uracile dans l'ARN. La différence entre ces deux bases est le remplacement d'un groupement méthyle en position 5 de la thymine par un hydrogène dans l'uracile. cette modification de structure ne modifie pas les propriétés d'appariement avec l'adénine[1], [2].
Stéréochimie
Sur le plan structural, la présence d'un oxygène sur la position 2'du ribose influence la conformation du cycle furanose du ribose. Ce cycle à cinq atomes n'est pas plan, ce qui conduit à deux conformères principaux du sucre, nommés C2'-endo ou C3'-endo. Dans l'ARN qui comporte un oxygène en position 2', la position C3'-endo est privilégiée[3], ce qui modifie profondément la structure des doubles hélices comportant des brins ARN. Ces duplex d'ARN forment une hélice de type A, différente de celle qui est observé de façon majoritaire dans l'ADN classique qui est une hélice de type B, où le désoxyribose est en conformation C2'-endo[4].
Double hélice d'ARN
L'hélice de type A, qu'adopte l'ARN quand il forme un duplex, a des propriétés géométriques assez différentes de celles de l'hélice de type B. Dans un premier temps le nombre de paires de bases par tour d'hélice est de 11 au lieu de 10 pour l'ADN en forme B. Le plan des paires de bases n'est plus perpendiculaire à l'axe de l'hélice, mais forme un axe d'environ 75 degrés avec ce dernier[7]. Il en résulte un déplacement de l'axe de l'hélice qui ne passe plus par le centre de l'appariement des bases, mais à l'intérieur du grand sillon. Ceci induit une augmentation du diamètre de l'hélice qui passe d'environ 20 Å pour l'ADN en forme B à à peu près 26 Å pour l'ARN en forme A[8]. Enfin la géométrie des deux sillons est profondément affectée : le petit sillon devient particulièrement accessible, alors que le grand sillon devient particulièrement profond, étroit et pincé. Ceci a un impact sur la manière dont l'ARN en duplex apparié peut interagir avec des protéines, car l'étroitesse du grand sillon est une barrière à l'accessibilité de ligands protéiques.
Structure in vivo
La plupart des ARN naturels sont présents sous forme simple brin dans la cellule, au contraire de l'ADN qui est sous forme d'un double-brin apparié. Les ARN simple brin se replient le plus fréquemment sur eux-mêmes, formant une structure intramoléculaire qui peut être particulièrement stable et particulièrement compacte. La base de cette structure est la formation d'appariements internes, entre bases complémentaires (A avec U, G avec C et , quelquefois, G avec U).
La description des appariements internes entre les bases d'un ARN se nomme la structure secondaire. Cette structure secondaire peut être complétée par des interactions à longue distance qui définissent alors une structure tridimensionnelle ou structure tertiaire.
La formation de la structure des ARN est fréquemment dépendante des conditions physico-chimiques environnantes et surtout de la présence de cations divalents, comme le magnésium Mg2+, dans la solution. Ces cations interagissent avec les groupements phosphate du squelette et stabilisent la structure, surtout en faisant écran à la répulsion électrostatique entre les charges négatives de ces phosphates. [9]
L'existence de structure tertiaire dans les ARN est à la base de la richesse de ses fonctions et surtout de sa capacité à catalyser des réactions chimiques (ribozymes).
Structure secondaire
La structure secondaire d'un ARN est la description de la totalité des appariements internes au sein d'une molécule simple brin[10]. Cet ensemble d'appariements induit une topologie spécifique, composée de région en hélice (tiges) et de régions non-appariées (boucles). Par extension, la structure secondaire recouvre aussi la description de cette topologie.
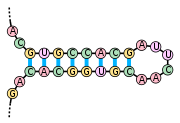
L'élément moteur de la formation de structures secondaires au sein d'un ARN simple-brin est l'existence de régions contenant des séquences répétées inversées, qui peuvent s'apparier pour former localement une structure en double hélice. A titre d'exemple, si l'ARN contient les deux séquences suivantes : --GUGCCACG----CGUGGCAC--, celles-ci forment un motif répété inversé, les nucléotides du second segment étant les complémentaires de ceux du premier, après inversion de leur sens de lecture. Ces deux segments peuvent par conséquent s'apparier de manière antiparallèle pour former une région localement en duplex. La région entre les deux segments forme alors une boucle reliant les deux brins du duplex. On parle alors de structure en tige et boucle ou en épingle à cheveux.
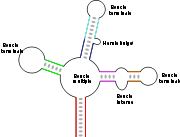
Dans des ARN de longueur plus importante, il peut exister des structures plus complexes, qui résultent de l'appariement de plusieurs régions complémentaires ou séquences répétées inversées. Selon la manière dont sont "emboîtées" ces différentes régions, on obtient des éléments topologiques variés, avec des tiges ou régions appariées, et divers types de boucles :
- Les boucles terminales, localisées à l'extrémité d'une tige.
- Les boucles internes, qui connectent deux tiges.
- Les boucles multiples, qui connectent trois tiges ou plus et forment des points de branchement de la structure.
- Les hernies (en anglais bulge) ou boucles latérales qui sont sur un seul des deux brins d'une hélice. La continuité de l'hélice n'est généralement pas affecté et la totalité des bases reste empilé de manière coaxiale, de part et d'autre de la hernie.
Il n'existe pas forcément une structure unique stable pour une séquence donnée et il arrive que certains ARN puissent adopter plusieurs conformations alternatives selon la liaison d'un ligand (protéine, petite molécule... ) ou des conditions physico-chimiques (force ionique, pH). On peut généralement suivre la formation ou la fusion de la structure secondaire d'un ARN par des mesures spectroscopiques. Ainsi par exemple, l'absorption dans l'UV des bases de l'ARN est plus importante à l'état déplié qu'à l'état replié (phénomène d'hyperchromicité).
Structure tertiaire
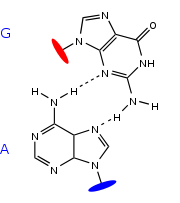
Appariements non-canoniques
Au delà de la topologie des boucles et des hélices composées de paires de bases standard, un ARN peut adopter une structure tridimensionnelle compacte, ou structure tertiaire, comme une protéine. A l'intérieur de cette structure, les hélices canoniques sont complétées par des appariements non canoniques, autrement dit différents des d'appariements de base classiques, de type Watson-Crick (A-U et G-C) et bancals (wobble, G-U). On a observé une grande variété de ces appariements dans les structures tridimensionnelles d'ARN résolues par cristallographie ou par RMN. On trouve par exemple des appariements Hoogsteen, des appariements "en cisaille" (sheared). Il existe aussi des interactions base-ribose.
Une nomenclature systématique de toutes ces interactions a été proposée par Eric Westhof et ses collaborateurs[11]. Plus de 150 types d'appariements ont été observés et ont été regroupés en douze grandes familles. Ces appariements non-canoniques impliquent toujours des liaisons hydrogènes entre les bases, qui sont coplanaires, comme dans les paires Watson-Crick.
Interactions à longue distance
Des appariements canoniques ou non-canoniques peuvent intervenir entre des régions distantes de la structure secondaire, fréquemment situées dans des boucles, ce qui sert à stabiliser un repliement compact de la structure.
Parmi ces interactions non canoniques à longue distance, on peut citer :
- Les pseudonœuds : Structure constituée par l'interaction d'une boucle avec une région localisée hors de la tige qui la délimite.
- Les triplex de brin : Surviennent quand une région simple brin vient s'insérer dans le grand sillon d'une région en hélice.
- Les interactions tétraboucle-récepteur : interaction entre boucles hyperstables de quatre nucléotides (tetraboucles) et structures en duplex ou quasi duplex.
Similitudes et différences entre l'ADN et ARN
- l'ARN a pour composant un sucre ribose à l'endroit où l'ADN a un désoxyribose ;
- la base uracile a un rôle pour l'ARN qui est rempli par la thymine dans l'ADN ;
- l'ARN est existe le plus souvent sous la forme d'un simple brin (monocaténaire), sauf chez quelques organismes tels les rétrovirus, alors que l'ADN est double brin (bicaténaire) avec une structure en double hélice ; L'appariement de base complémentaire dans un brin d'ADN peuvent lui conférer des propriétés d'une structure double brin.
- l'ARN est court : de quelques dizaines à quelques milliers de nucléotides, contre quelques millions à quelques milliards dans l'ADN.
Les trois premières différences donnent à l'ARN une stabilité bien moindre que celle de l'ADN :
- les deux fonctions alcools géminés du ribose sont susceptibles d'hydrolyse alcaline et acide alors que le désoxyribose de l'ADN, qui a perdu ce groupe fonctionnel y est insensible (D de ADN veut dire Désoxyribonucléique, i. e. ayant perdu une fonction oxy (-OH), au niveau 2'du ribose)
Le ribose possède un groupement hydroxyle en position 2', qui est absent dans le désoxyribose de l'ADN. Cette fonction 2'-OH a des incidences multiples sur la structure de l'ARN. Dans un premier temps sur le plan chimique, cette fonction alcool rend l'ARN sensible à l'hydrolyse alcaline. La présence des deux oxygènes en cis sur le positions 2'et 3'rend envisageable la cyclisation du phosphate sur les positions 2'et 3', qui se produit particulièrement rapidement quand une base vient arracher le proton du 2'-OH. Cette cyclisation du nucléotide provoque une coupure de la chaîne ribose-phosphate et libère des extrémités 5'-OH et 2', 3'phosphate cyclique[12].
- L'uracile est moins ‘‘coûteux''à produire pour les organismes vivants que la thymine, mais se convertit lentement en cytosine. Ce serait pour cette raison que l'ARN a de l'uracile et l'ADN de la thymine : la cellule produit énormément d'ARN mais ne le conserve pas, c'est par conséquent le coût de production qui prime sur la stabilité de l'information. À l'inverse, l'ADN doit conserver une information longtemps mais n'est produit que rarement (lors de la division cellulaire), c'est par conséquent la stabilité qui prime sur le coût.
- si un brin d'ARN, monocaténaire, est endommagé, il se produira une erreur irrécupérable ; par contre, si un des deux brins d'ADN est endommagé, la cellule peut utiliser le brin intact pour réparer.
D'un point de vue évolutif, certains éléments permettent de penser que l'ARN serait antérieur à l'ADN comme support de l'information génétique, ce qui expliquerait ses fonctions plus étendues et sa généralisation. L'ADN serait apparu plus tard et n'aurait supplanté l'ARN que pour le rôle de stockage à long terme, à cause de sa plus grande stabilité.
Fonction de l'ARN dans la cellule
Les ARN sont impliqués dans différents aspect de la synthèse des protéines :
Dans une cellule eucaryote, quatre catégories principales d'ARN sont présentes : l'ARn messager, l'ARN de transfert, l'ARN ribosomique et l'ARN nucléaire. [13]
Dans les cellules, les ARN remplissent trois rôles différents et complémentaires :
- Support temporaire de l'information génétique. C'est l'ARN messager qui remplit ce rôle, il est utilisé par la cellule pour transmettre l'information correspondant à un gène donné hors du noyau, puis pour synthétiser des protéines à partir de ces informations.
- Catalyseur enzymatique. Comme les protéines, les ARN peuvent se replier en trois dimensions pour former des structures complexes. Ces structures autorisent certains ARN de se comporter comme des enzymes, on parle alors de ribozyme. Le ribosome, la ribonucléase P et certains introns sont des ribozymes. On pense que la machinerie d'épissage des ARN messagers (le spliceosome), est aussi aussi un ribozyme, même si la démonstration formelle n'en a pas toujours été apportée.
- Guide pour des enzymes. Certains ARN sont utilisés comme co-facteurs par des protéines pour permettre leur ciblage vers des séquences spécifiques. Parmi ceux-ci, on peut citer les petits ARN nucléolaires (snoARN), qui guident les enzymes de modification de l'ARN ribosomique, l'ARN télomérique, qui est un cofacteur de la télomérase, l'enzyme qui produit les extrémités des chromosomes, ou encore les ARN interférents.
L'ARN est par conséquent une molécule particulièrement polyvalente, ce qui a conduit Walter Gilbert, co-inventeur du séquençage de l'ADN à proposer en 1986 une hypothèse selon laquelle l'ARN serait la plus ancienne de l'ensemble des macromolécules biologiques[14]. Cette théorie, dite du "RNA World" (le monde de l'ARN), sert à s'affranchir d'un Paradoxe de l'œuf et de la poule qui survient quand on cherche à savoir qui des protéines (catalyseurs) et de l'ADN (information génétique) sont apparus en premier. L'ARN, capable de combiner à la fois les deux types de fonctions serait le précurseur universel.
Synthèse de l'ARN à partir de l'ADN
La synthèse d'une molécule d'ARN à partir de l'ADN est un processus complexe qui fait intervenir une enzyme de la famille des ARN polymérase mais aussi des protéines associées. Les différentes étapes de cette synthèse sont l'initiation, la propagation et la terminaison. Les ARN synthétisées dans les organismes procaryotes sont différentes des ARN des cellules de mammifères. [13]
La terminaison
Les expériences de Taylor (1957) et de Meselson et Stahl (1958) ont permis de démontrer que la réplication s'effectue par un mécanisme semi-conservatif. Les deux autres hypothèses pour la réplication impliquaient des modèles conservatif ou dispersif. La réplication est une réaction qui s'effectue seulement dans le sens 5'à 3'.
Références
- ↑ Sænger, W (1984). Principles of nucleic acid structure. Springer. ISBN 0387907629.
- ↑ Barciszewski J, Frederic B, Clark C (1999). RNA biochemistry and biotechnology. Springer, 73–87. ISBN 0792358627. OCLC 52403776.
- ↑ Sudaralingam M., «Stereochemistry of nucleic acids and their forments. IV. Allowed and preferred conformations of nucleosides, nucleoside mono-, di-, tri-, tetraphosphates, nucleic acids and polynucleotides», dans Biopolymers, vol. 7, 1969, p. 821-860
- ↑ Langridge R., Gomatos P. J., «The Structure of RNA. Reovirus RNA and transfer RNA have similar three-dimensional structures, which differ from DNA. », dans Science, vol. 141, 1963, p. 694-698 [texte sur PMID]
- ↑ Drew H. R., Wing R. M., Takano T., Broka C., Tanaka S., Itakura K., Dickerson R. E., «Structure of a B-DNA dodecamer : conformation and dynamics. », dans Proc. Natl. Acad. Sci. USA, vol. 78, 1981, p. 2179-2183 [texte sur PMID]
- ↑ Klosterman P. S., Shah S. A., Steitz T. A., «Crystal structures of two plasmid copy control related RNA duplexes : An 18 base pair duplex at 1.20 A resolution and a 19 base pair duplex at 1.55 A resolution. », dans Biochemistry, vol. 38, 1999, p. 14784-14792 [texte sur PMID]
- ↑ Rosenberg J. M., Seeman N. C., Kim J. J., Suddath F. L., Nicholas H. B., Rich A., «Double helix at atomic resolution. », dans Nature, vol. 243, 1973, p. 150-154 [texte sur PMID]
Day R. O., Seeman N. C., Rosenberg J. M., Rich A., «A Crystalline Fragment of the Double Helix : The Structure of the Dinucleoside Phosphate Guanylyl-3', 5'-Cytidine. », dans Pro. Natl. Acad. Sci. USA, vol. 70, 1973, p. 849-853 [texte sur PMID] - ↑ Rich A., Davies D. R., «A new two stranded helical structure : Polyadenylic acid and polyuridylic acid. », dans J. Am. Chem. Soc. , vol. 78, 1956, p. 3548-3549 [lien DOI]
- ↑ Woodson S. A., «Metal ions and RNA folding : a highly charged topic with a dynamic future. », dans Curr. Opin. Chem. Biol. , vol. 9, 2005, p. 104-109 [texte sur PMID]
- ↑ Doty P., Bœdtker H., Fresco J. R., Haselkorn R., Litt M., «Secondary Structure in Ribonucleic Acids», dans Proc. Natl. Acad. Sci. USA, vol. 45, 1959, p. 482-499
- ↑ Leontis N. B., Stombaugh J., Westhof E., «The non-Watson-Crick base pairs and their associated isostericity matrices. », dans Nucleic Acids Res. , vol. 30, 2002, p. 3497-3531 [texte sur PMID]
- ↑ Markham R., Smith J. D., «The Structure of Ribonucleic Acids 1. Cyclic nucleotides produced by ribonuclease and by alkaline hydrolysis», dans Biochem. J. , vol. 52, 1952, p. 552-557 [texte sur PMID]
- ↑ a b Biochimie de Harper, Harold A Harper, A Harold
- ↑ Walter Gilbert, The RNA World Nature 319 (1986) : 618
Voir aussi
- Double-chaîne d'ARN complémentaire introduite artificiellement dans la cellule ou codée naturellement par des régions de génome traversées par des événements de transcription contraires. Induit l'inactivation de gènes (Interférence par l'ARN) par un processus de dégradation de l'ARN messager de séquence correspondante.
- ARN partiellement double-brin ayant une structure tige-bouche en «épingle à cheveux». Codé par le génome le microARN est maturé en un très petit ARN de 21-24 bases et s'apparie avec l'extrémité 3' d'un ou de plusieurs ARN messagers cibles. Cet appariement induit une inhibition de la traduction.
- ARN qui permet aux cellules de lutter contre certains envahisseurs, comme les virus. Ce mécanisme interrompt le travail de l'ARN messager qui transporte le code génétique indispensable à la synthèse d'une protéine du noyau de la cellule vers le site de fabrication de la protéine.
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 17/03/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité