Marqueur de séquence exprimée
Un marqueur de séquence exprimée, ou Expressed Sequence Tag, est une courte portion séquencée d'un ADN complémentaire, utilisée comme marqueur pour différencier les gènes entre eux dans une séquence ADN et identifier les gènes homologues dans d'autres espèces.
Recherche sur Google Images :
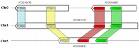
Source image : ead.univ-angers.fr Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Page(s) en rapport avec ce sujet :
- Le séquençage systématique de marqueurs de séquence exprimée (MSE) peut servir de base à la découverte de gènes associés à l'infection, à la réplication et ... (source : books.google)
- Vous pourriez rechercher une correspondance avec un marqueur de séquence exprimé (EST).... 7. En plus des séquences codantes, votre génome renferme. a. de l'ADN non codant au sein des gènes (par exemple des introns).... (source : ravenbiologie)
- Par exemple le STS (sequence -tagged site) est un marqueur de choix pour cette... -Pour connaître la séquence des ADN, on fait synthétiser un brin d'ADN par une... de ses extrémités : on obtient les EST (marqueurs de séquences exprimées).... La comparaison des séquences du gène chez les individus sains et les ... (source : genet.univ-tours)
Un marqueur de séquence exprimée, ou Expressed Sequence Tag (EST), est une courte portion séquencée d'un ADN complémentaire (ADNc), utilisée comme marqueur pour différencier les gènes entre eux dans une séquence ADN et identifier les gènes homologues dans d'autres espèces[1].
Parce qu'il est le plus souvent assez facile de récupérer des brins d'ARNm des cellules, les biologistes récupèrent ces séquences et les convertissent en ADNc, qui est énormément plus stable. Un ARNm étant nécessairement l'expression d'un gène du génome, cet ADNc n'est pas une copie exacte de la séquence ADN qui a généré l'ARN car, suite à l'épissage, l'ARN ne garde pas les régions non codantes de l'ADN (introns).
Si le génome est correctement identifié, les gènes le composant ne le sont pas toujours tous. Et certains gènes identifiés ont une fonction toujours inconnue. En utilisant les EST, on cherche à identifier un gène par le chemin inverse, c'est-à-dire qu'on prend la séquence d'ARN pour trouver la séquence d'ADN qui l'a créée. Cela va permettre de localiser le gène dans le génome, tout en n'ayant aucune information sur le gène lui-même.
Pour effectuer une recherche sur la séquence d'ADN originelle, on n'utilise qu'une partie de l'information contenue dans l'ADNc, une centaine de nucléotides. Cette sous-séquence est prise en début ou en fin de séquence. Le début est assez bien conservé d'une espèce à l'autre ; aussi, pour identifier de manière unique le gène en cause, on a tendance à utiliser la fin de la séquence comme base de recherche.
Notes et références
- ↑ Ghislaine Cleret de Langavant, Bioéthique : méthode et complexité [lire en ligne], pp. 241-242, PUQ, 2001, 309 p. (ISBN 2760511065) .
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 17/03/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité