Rétrotransposon
Les rétrotransposons appartiennent à la grande famille des éléments transposables. Ils correspondent à des séquences d'ADN endogènes capables de se déplacer et en particulier de se multiplier dans le génome de l'hôte.
Recherche sur Google Images :
Source image : en.wikipedia.org Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Page(s) en rapport avec ce sujet :
- Les éléments transposables, et surtout les rétrotransposons, ..... profil d'insertion si l'amorce rétrotransposon est choisie dans la LTR...... Rapid isolation of plant Ty1-copia group retrotransposon LTR sequences for molecular... (source : legs.cnrs-gif)
- Une copie du code ADN du rétrotransposon est faite en une molécule d'ARN, qui..... sentant de potentiels LTR, pour former un élément entier de 1000 à ... (source : ftp.irisa)
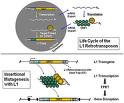
Les rétrotransposons appartiennent à la grande famille des éléments transposables (éléments de Classe I à intermédiaire ARN). Ils correspondent à des séquences d'ADN endogènes capables de se déplacer et en particulier de se multiplier dans le génome de l'hôte. Ils se différencient des transposons (Classe II des éléments transposables) par leur intermédiaire à ARN (et non ADN). Le préfixe rétro- vient du fait que les rétrotransposons vont "à l'inverse" du dogme central de l'ADN, car leur ARN est "rétro"-transcrit en ADN. Certains d'entre eux, les rétrotransposons à LTR (Long Terminal Repeat sequence), sont apparentés aux rétrovirus, mais sans cependant être infectieux.
Classification
La Classe des rétrotransposons est particulièrement important en nombre et en "qualité". Fondée à la fois sur des différences structurales, et sur la présence ou l'absence de longues régions terminales répétées (LTR), nous distinguons deux classes de rétrotransposons : les rétrotransposons à LTR ou sans LTR. Ces deux classes d'éléments changent aussi par leur mécanisme d'intégration.
- Ceux possédant des LTR vont synthétiser un ADN complémentaire (ADNc) à partir de leur ARNm (reverse transcription) dans des particules pseudovirales (supposées strictement cytoplasmiques), puis intégrer cet ADNc à un nouveau locus chromosomique.
- Ceux n'ayant pas de LTR vont synthétiser cet ADNc directement au site cible d'intégration, provoquant quelquefois eux-mêmes la coupure du double brin d'ADN indispensable à cette intégration.
Les rétrotransposons à LTR
Ces éléments ressemblent par leur cycle de réplication et leur structure aux rétrovirus. Comme ces derniers, les rétrotransposons à LTR actifs codent essentiellement et le plus souvent deux protéines de type gag et pol. Cependant, les rétrotransposons ont un cycle seulement intracellulaire, c'est-à-dire qu'ils ne peuvent pas infecter la cellule voisine, comme le font les rétrovirus. Cependant, des particules pseudo-virales de l'élément gypsy de Drosophila melonogaster, purifiées sur gradients de sucrose, sont capables d'infecter des cellules mises à leur contact. cette capacité infectieuse est donnée par la présence d'une 3e protéine majeure, codant l'enveloppe. Cette enveloppe est théoriquement présentes chez certains rétrotransposons à LTR, proche de gypsy. Qui plus est , les rétrotransposons sont retrouvés chez l'ensemble des eucaryotes (ensemble des organismes composés de cellules à noyau, par opposition aux procaryotes) tandis que les rétrovirus n'ont été trouvés à l'heure actuelle que chez les animaux (petite partie des eucaryotes).
Les rétrotransposons à LTR sont le plus souvent divisés eux-mêmes en quatre groupes : Ty1 /copia, Ty3 /gypsy, DIRS et BEL. On y adjoint quelquefois les LARDs et les TRIMs chez les végétaux, où les DIRS et les BEL sont absents. Il y a une grande variation au sein de ces groupes :
- organisation différentes des phases de lecture, du type de protéine, et des capacités codantes selon l'élément
- localisation du Primer Binding Site (PBS) où s'amorce la
- choix de l'amorce pour la (un ARNt entier, partiel ou pas d'amorce du tout)
- distance de fixation de cette amorce en aval du LTR
- ou encore longueur de la duplication du site d'insertion.
Les rétrotransposons non-LTR
Les éléments appartenant à cette classe possèdent et utilisent aussi une. Leur cycle de réplication diffère de celui des rétrotransposons. La synthèse d'ADNc n'a pas lieu dans le cytoplasme de la cellule, mais directement au site d'insertion de la nouvelle copie : un brin d'ADN du site cible serait clivé par l'endonucléase de l'élément, l'ARNm y est rattaché, donnant la possibilité la transcription inverse d'un brin. L'ARNm est ensuite retiré, laissant la place pour la synthèse du deuxième brin d'ADN. Ce mécanisme est nommé Target-Primed Reverse Transcriptase (ou TPRT). Les rétrotransposons sans LTR sont divisés en cinq groupes : R2, L1, RTE, I et Jockey. Leur phylogénie est complexe car cette classe regroupe la plupart d'éléments.
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 17/03/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.


 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité