Acide ribonucléique messager
L'acide ribonucléique messager, ou ARN messager ou ARNm, est une copie de l'ADN utilisée comme intermédiaire par les cellules dans la synthèse des protéines.
Recherche sur Google Images :
Source image : tsspesvt.over-blog.com Cette image est un résultat de recherche de Google Image. Elle est peut-être réduite par rapport à l'originale et/ou protégée par des droits d'auteur. |
Page(s) en rapport avec ce sujet :
- acide ribonucléique (A. R. N. ). Documents associés... du cytoplasme qui permettent le déchiffrage du code génétique inscrit dans l'A. R. N. messager.... (source : larousse)
- ADN (acide désoxyribonucléique) et ARN (acide ribonucléique).... séquence spécifique de bases, le codon, de l'ARN messager lors de la synthèse protéique.... (source : ulaval)
- ARN (Acide ribonucléique). Dans les cellules, on distingue plusieurs types d'ARN suivant leur fonction. Les trois types principaux sont : les ARN messagers, ... (source : interstices)
| Acides nucléiques |
|---|
L'acide ribonucléique messager, ou ARN messager ou ARNm, est une copie de l'ADN utilisée comme intermédiaire par les cellules dans la synthèse des protéines.
Principes
L'information génétique d'une cellule eucaryote est contenue dans son noyau sous forme de molécules d'ADN (les procaryotes ne possèdent pas de noyau). C'est cette information génétique qui va être utilisée comme guide pour fabriquer les chaînes polypeptidiques que sont les protéines. Cependant la synthèse des protéines se déroule dans le cytoplasme de la cellule. De multiples communications ont par conséquent lieu entre noyau et cytoplasme, dans le cadre de l'expression de l'information génétique. L'ARN messager y a un rôle central : c'est le vecteur de l'information génétique.
Synthétisé à partir de l'ADN, il en transcrit l'information ; puis, après transport dans le cytosol (chez les eucaryotes toujours), cet ARNm est lu par de nombreux ribosomes. Ceux-ci accèdent ainsi indirectement à l'information génétique.
L'interface entre l'ARN messager et la protéine est surtout assurée par les ARN de transfert (ARNt ou tRNA).
Bien que le problème ne se pose pas dans les mêmes termes chez les procaryotes (pas de compartimentation empêchant a priori le passage direct de l'ADN aux protéines), il y a également passage par un intermédiaire ARNm dans l'expression de l'information génétique chez ces derniers. L'ARNm est par conséquent une molécule universelle dans le monde vivant.
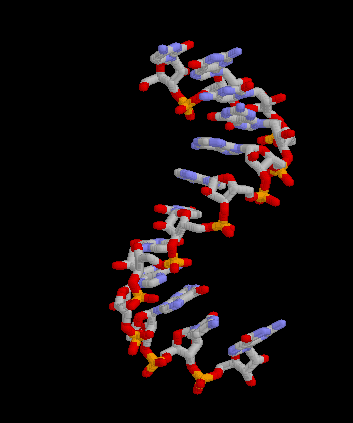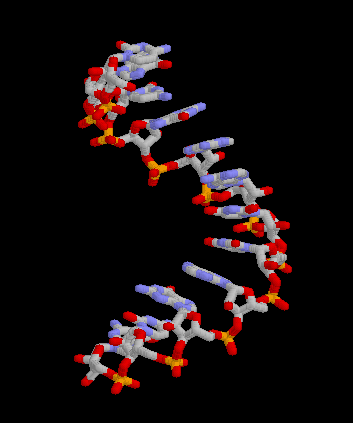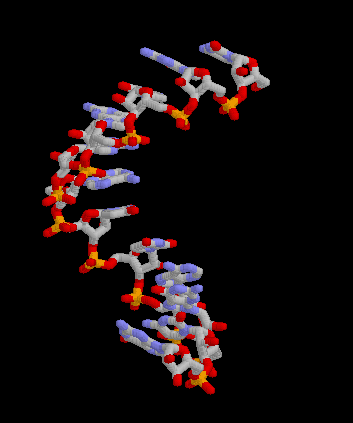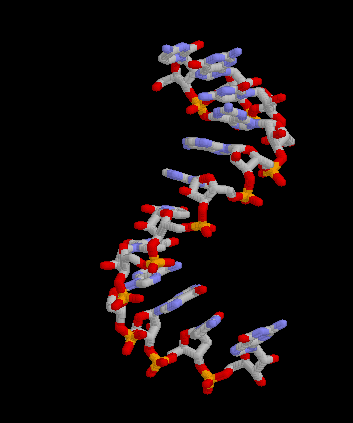
Structure
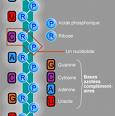
L'ARNm est un acide nucléique résultant de la polymérisation de ribonucléotides reliés par des liaisons phosphates. Comme le terme "ARN" l'indique, l'ose (ou plus familièrement "sucre") présent dans les ribonucléotides est le ribose. Les bases azotées présentes sur les ribonucléotides sont l'Adénine et l'Uracile (complémentaires entre elles) mais aussi la Guanine et la Cytosine (complémentaires de même). L'ARNm emploie par conséquent la base U, contre la base T dans l'ADN. Pour une hypothèse explicative de cette différence, voir l'article sur l'Uracile. Au contraire de l'ADN génomique (en général), l'ARNm est une molécule monocaténaire, c'est-à-dire constituée d'un seul brin. Ceci n'empêche pas des repliements (quelquefois complexes et particulièrement structurés) de la molécule sur elle-même faisant intervenir la complémentarité entre bases azotées.
Phase de
L'ARNm consiste en une copie sélective d'un fragment d'ADN (gène de structure). L'opération de copie nommée transcription se déroule dans le noyau de la cellule chez les Eucaryotes et dans le cytoplasme chez les Procaryotes, le matériel génétique y résidant. La transcription a aussi lieu dans les organites semi-autonomes comme les mitochondries et les chloroplastes qui contiennent une partie du matériel génétique qui leur est indispensable. Elle s'effectue en présence d'enzymes spécifiques nommées ARN polymérases et des nucléotides nécessaires à la synthèse de l'ARN.
Les ARN polymérases écartent les deux brins qui forment la molécule d'ADN puis synthétise la molécule d'ARN en se servant du brin dit transcrit comme matrice. À la fin de cette transcription, deux séquences complémentaires sont transcrites, ce qui entraîne la formation d'un terminateur donnant la possibilité le détachement de la molécule d'ARN. Chez les Procaryotes, la molécule d'ARNm est directement transcriptible. Chez les Eucaryotes (noyau et organites), la molécule synthétisée par les ARN polymérases est dans de nombreux cas de l'ARN prémessager qui doit subir une maturation post-transcriptionnelle dans le noyau avant de pouvoir être traduit. En effet, les séquences codantes (Exons) chez les eucaryotes sont interrompues par des séquences non codantes (Introns). Au cours de la maturation post-transcriptionnelle aura surtout lieu le phénomène d'épissage assurant le rassemblement des séquences résultant de la transcription des exons tandis que les séquences résultant de la transcription des introns seront retirées de l'ARN prémessager.
Phase de maturation des transcrits ou modification post-transcriptionnelle
Après de l'ADN en ARN, on obtient une structure moléculaire qui n'est pas toujours "mature", et qui ne peut pas, en l'état, être directement à l'origine d'une protéine. Ce manque de maturité vient du fait qu'on distingue dans sa structure des introns et des exons, seuls les exons sont utiles pour la synthèse de la protéine et les introns doivent être retirés avant que la phase de traduction puisse s'effectuer. Lors de cette maturation, il faudra aussi transformer un peu cet acide ribonucléique pour le rendre plus résistant face à son environnement. L'objectif de cette maturation est de transformer cet ARN pré-messager (ARNpré-m) en ARN messager (ARNm).
- Les ribonucléoprotéines nucléaires hétérogènes
Après synthèse, le préARNm est entouré de protéines A, B et C nucléaires, la formation d'un complexe tétramérique l'ensemble des 600 nucléotides sert à maintenir les ARNpré-m sous forme monobrin et ainsi permettre la suite de la maturation.
- Les petits ARN nucléaires hétérogènes (ARNsn)
Ces ARNsn (small nuclear) sont transcrits de l'ADN par l'ARN polymérase III, ils sont composés en majorité d'Uracile. Ils vont former un complexe avec 8 petites protéines basiques nucléaires. Le tout forme les ribonucléoprotéines small nuclear (RNPsn), qui sont de petites protéines ribonucléiques (notée de U1 à U6)
Le rôle de ces protéines intervient au niveau de l'épissage des ARNpre-m mais aussi dans la méthylation de l'ARN pré ribosomique du nucléole (ARNpré-r).
- Modifications en 5'
Ces modifications se font au début de la synthèse d'ARNm (après 30 nucléotides). Toutes ces modifications se font en majorité par voie enzymatique.
- La première modification en 5'est l'ajout d'une coiffe à l'ARN cette coiffe est constituée de 7 méthyl-guanines (7mG). Cet ajout se fait par une enzyme, la guanylméthyl-transférase. Il y aura la formation d'un pont 5'-5'triphosphate dans le sens opposé de la synthèse normale. Tout ceci a pour but de faire barrière à l'action des 5'exonucléase, par conséquent de protéger l'ARN de son environnement mais également un rôle dans l'épissage, dans le transport transnucléaire mais aussi dans la traduction.
- La seconde est une méthylation des riboses en 2'-OH.
- Enfin la troisième modification est la formation de N6-méthyl-adénosine, par l'action d'une ARN méthylase qui est cependant différente de l'ADN méthylase de maintenance.
- Modifications en 3'
Cette modification est une polyadénylation, c'est à dire, il va y avoir ajout en 3'de l'ARN d'une queue de poly (A). Séquence de cette queue :
- séquence signal : 5'-AAUAAA-3'
- site d'hydrolyse : 5'-YA-3'où Y est une base pyrimidique (U ou C)
- région riche en -GU-
La séquence totale est par conséquent de la forme : 5'-AAUAAA-.......... -YA-.......... -GU-5'
Il va ensuite y avoir l'intervention de l'ARN poly (A) polymérase qui va ajouter 250 résidus adényliques à cette queue. Cette queue a pour rôle la protection contre les ARNases et nucléolases hydrolysant en 3'-OH, ainsi qu'un rôle de stabilisation en vue de la traduction.
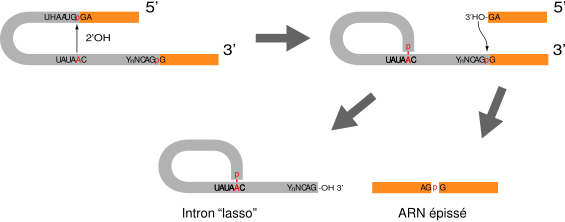
L'épissage
L'épissage est un procédé de maturation des ARNpré-m se déroulant dans le nucléoplasme et facilitant l'excision des introns et la mise en continuité de différents exons. On peut par conséquent remarquer que les gènes sont morcelés en : Exons : séquence traduite en protéine et en Introns qui ne seront pas traduits en protéine mais retiré lors de l'épissage.
Séquences remarquables des introns :
- en 5', GU (1)
- en 3', poly (Y) 11NC-AG (1et2)
- Entre 3'et 5', une séquence -CURAY- de branchement à 30 nucléotides de 3'
On utilise ici les codes à une lettre pour les combinaisons de bases pour représenter les variations envisageables de bases.
Principe de l'épissage :
Il y a dans un premier temps une hydrolyse en amont de G en 5'de l'intron, puis formation d'une liaison 2'5'phosphodiester de cette guanine avec l'Adénine de la séquence -CURAY-, ce qui conduit à une boucle intronique, couramment nommé le lasso. On se retrouve alors avec un exon qui a son extrémité 3'OH libre. Il va ensuite y avoir une hydrolyse de la liaison phosphodiester en aval de G en 3', ce qui conduit à l'expulsion du lasso, les exons se retrouvent alors libres et vont être joints par une liaison phosphodiester.
Détail du mécanisme
Il y a intervention des snRNP dont la partie ARN s'apparie à la partie terminale des introns (commune pour l'ensemble des introns), la partie terminale de U1 se lie au site 5'-GU- et la partie terminale de U2 se lie au point de branchement A du -CURAY-. Puis, on assiste à une fixation de U4, U5, U6 sur U2 ce qui entraîne la formation du lasso
Phase de Traduction
Les ARNm sont ensuite traduits en protéines par l'intermédiaire des ribosomes dans le cytoplasme. Les deux sous-unités (petite sous-unité 40S et grande 60S) des ribosomes s'assembleront sur une séquence leader de la molécule d'ARNm.
4 étapes de la traduction :
1. liaison entre ribosome et ARNm --> étapes de régulation
2. initiation de la traduction --> étape de régulation
3. élongation (synthèse de la chaîne peptidique)
4. terminaison (relâchement de la chaîne)
Le ribosome contient 3 sites fixant l'ARN :
site A : aminoacyl-ARNt
site P : peptidyl-ARNt
site S : sortie
Ces sites ne sont jamais occupés en même temps!
ARNm eucaryote est monocistronique : le ribosome rentre via la coiffe 5' (unique aux eucaryotes)
ARNm procaryote est polycistronique : il y a plusieurs sites de liaisons au ribosome.
ARN de transfert :
C'est une molécule dont la forme 2D rappelle une forme de feuille de trèfle. La forme *D se rapproche d'un L. L'ARNt possède un anticodon qui s'appareillera lors de la traduction à un codon homologue sur l'ARNm correspondant à un acide aminé. En effet l'acide aminé est indirectement choisi par son codon :
1. liaison (covalente) acide aminé à ARNt
2. l'ARNt se lie à son codon sur l'ARNm
(Rappel : 1 codon = 3 nucléotides = 1 acide aminé)
Il est important que l'acide aminé soit lié au bon ARNt ! Fidélité est principale : taux d'erreur < 0.01%.
Les synthétases sont par conséquent les gardiens du code :
Discrimination : voient la différence de charges quand qu'il y a des acides aminés polaires et chargés, cela représente une différence d'énergie importante entre les 2 acides aminés. Le problème se pose quand il y a 2 acides aminés non polaires. À ce moment on regarde les contacts de Van der Waals mais il y a 1% d'erreur, cela est trop grand. Les synthétases ont une fonction de relecture : ARNt sur un site de la prot, un acide aminé sur un autre site, il y a 1 erreur/100 --> transfert du mauvais acide aminé sur un autre site (de nouveau 1/100) : 1/100 * 1/100 = 1/10000 = 0.01%.
Codage de l'information sur l'ARNm
- Initiation
Fixation d'ARN polymérase sur la séquence promoteur.
- Élongation
Synthèse d'ARN.
- Terminaison
La séquence de terminaison à la fin du gène arrête la transcription. L'ARN est libéré.
Voir aussi
Lien externe
Recherche sur Amazone (livres) : |
Voir la liste des contributeurs.
La version présentée ici à été extraite depuis cette source le 17/03/2009.
Ce texte est disponible sous les termes de la licence de documentation libre GNU (GFDL).
La liste des définitions proposées en tête de page est une sélection parmi les résultats obtenus à l'aide de la commande "define:" de Google.
Cette page fait partie du projet Wikibis.



 Accueil
Accueil Recherche
Recherche Début page
Début page Contact
Contact Imprimer
Imprimer Accessibilité
Accessibilité